V LA REGULATION DE LA SYNTHESE DES PROTEINESLa régulation de la synthèse protéique est une
absolue nécessité aussi bien chez les procaryotes que chez les eucaryotes.
IV.1 Chez les Procaryotes Chez les procaryotes, le contrôle de l’expression des gènes permet
d’adapter la cellule d’une part à ses besoins nutritionnels et
à son
environnement, mais aussi de lui assurer une croissance et une division
cellulaire normales. Il faut adapter la synthèse des protéines en
fonction des besoins ( coût énergétique élevé).
IV.1.1 Régulation au niveau de la transcription.Il existe ainsi deux types de régulations :
1) Par induction -------------> plus de synthèse.
2) Par répression------------> moins de synthèse.
IV.1.1.1 L’induction Exemple de l’Opéron lactose
-
Bases expérimentales- On sait que le lactose est hydrolysé en glucose + galactose par une enzyme nommée
β galactosidase .
Colonie de Colibacilles :
Si placée dans une solution de glucose, on ne trouve pas de β galactosidase dans le milieu de culture.
Repiquage d’une fraction de la colonie dans un milieu contenant du lactose : On détecte une présence abondante de
β galactosidase dans le milieu de culture.
Repiquage à nouveau d’une fraction de cette dernière colonie dans un
milieu ne contenant pas de lactose, mais du glucose.On ne trouve plus de
β galactosidase
Conclusion : La synthése de β galactosidase est induite par la présence de lactose.
Explication : En fait l’utilisation du lactose nécessite la présence de
trois enzymes.
La β-galactosidase (gène
lacZ). Elle hydrolyse la liaison β1-4 osidique des β-galactosides.
La lactose perméase (gène
lacY). Cette protéine membranaire permet l’entrée du lactose dans la cellule.
La thiogalactoside transacétylase (gène
lacA).
Son rôle n’est pas bien connu. Elle acétyle les β-galactosides non
métabolisables qui peuvent alors être éliminés hors de la cellule par
diffusion à travers la membrane plasmique.
Le lactose est donc l’inducteur de trois enzymes.
Dans
l’opéron lactose, on trouve les trois gènes correspondant aux trois enzymes indispensables à la dégradation du lactose.
Il est constitué de :
1.trois gènes de structure ( lac Y, lac A , Lac Z ).
2.un opérateur
3.un promoteur chevauchant l’opérateur
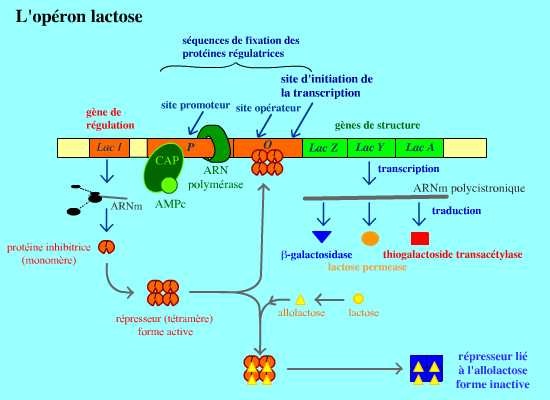
Ces
trois gènes de structure sont précédés par une région responsable de la
régulation de leur expression. Cette région régulatrice comprend le promoteur et l’opérateur. On trouve également en amont de l’opéron lactose, le gène régulateur (
lacI)
qui code une protéine régulatrice. Celle-ci agit en
inhibant l'expression des gènes de l'opéron lactose par
transactivation en se liant spécifiquement sur l’ADN au niveau de
l’opérateur. L'expression de ce répresseur est
constitutive, c'est à dire qu'il est exprimé quelque soient les
conditions de croissance de la bactérie. Par contre, son affinité pour
l'opérateur sera modifiée en présence
de lactose.
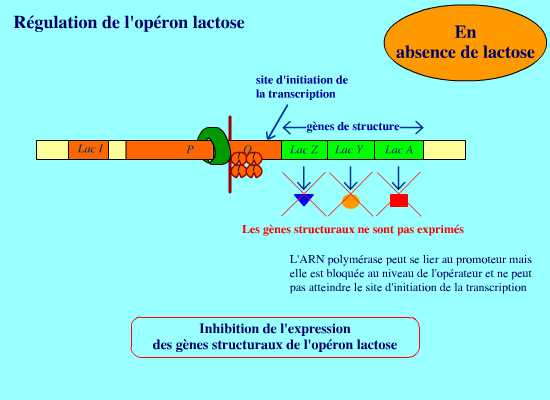
 Fonctionnement de l’opéron lactoseEn présence de glucose ( pas de lactose ), inhibition de la transcription, les trois enzymes n’apparaissent pas.
Fonctionnement de l’opéron lactoseEn présence de glucose ( pas de lactose ), inhibition de la transcription, les trois enzymes n’apparaissent pas.Les trois gènes de structure sont donc muets, ils sont répressés, réprimés.
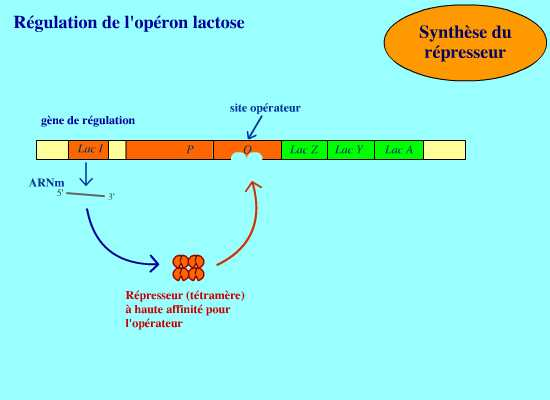
Le mécanisme de cette répression est le suivant :
Le gène régulateur possède son propre promoteur qui est distinct de
celui des gènes de structure. Ce gène code pour une protéine:
le répresseur.
Le répresseur est un tétramère de sous-unités identiques de 38 000
daltons chacune. Il possède une haute affinité pour l’opérateur. Dans
ces conditions,
si le répresseur est fixé sur l’opérateur, l’ARN polymérase ne peut pas transcrire
les gènes de structure car elle ne peut pas progresser vers les gènes de structure à partir de son site de fixation qui est le promoteur.En présence du lactose, Levée de l'inhibition de la transcription.Nécessité pour la bactérie de synthétiser trois enzymes pour
survivre, la répression est levée, les gènes sont exprimés , il y a
dérépression .
Le mécanisme de la dérépression est le suivant :
l y a
dérépression par le lactose. Le répresseur synthétisé
par le gène régulateur est reconnu par le lactose et s’associe avec lui.
Il y a impossibilité de fixation du répresseur sur l’opérateur. Si le
répresseur est déjà fixé sur l’opérateur, il est décroché par
l’inducteur (allolactose).
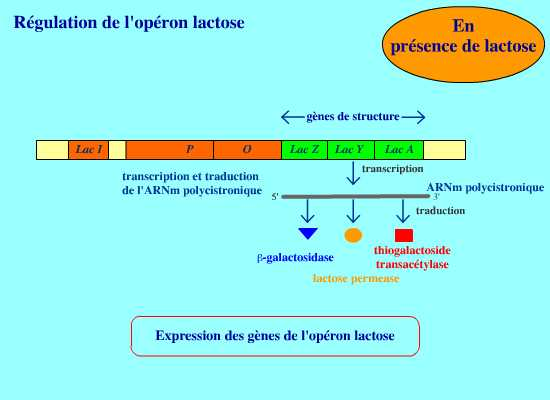
Dans ces conditions, l’ARN polymérase peut librement transcrire les
trois gènes de structure puisqu’elle peut se fixer sur le promoteur. Il
est important de comprendre que les trois gènes de structure
appartiennent à
un système polycistronique. Le mARN formé lors de la transcription code pour les trois protéines (perméase, transacétylase et b-galactosidase).
 Présence de Glucose et de Lactose , répression catabolique, régulation positive.Cultivé
Présence de Glucose et de Lactose , répression catabolique, régulation positive.Cultivé
en présence de lactose et de glucose comme source de carbone, E. coli
métabolise d’abord le glucose. La bactérie arrête ensuite temporairement
sa croissance jusqu’à que les gènes de l’opéron lactose subissent
l’induction qui permettra le métabolisme du lactose. Même si le lactose
est présent depuis le début de la croissance bactérienne, la cellule ne
commence pas l’induction des enzymes nécessaires au catabolisme du
lactose avant que tout le glucose présent ne soit utilisé.Initialement,
ce phénomène a été attribué à la répression de l’opéron lactose par un
catabolite du métabolisme du glucose. On parle de répression par un
catabolite d’où le nom donné à la protéine CAP (" catabolite gene
activator protein " ou protéine activatrice des gènes soumis à la
répression par un catabolite). L’opéron lactose n’est pas le seul opéron
dans E.coli sensible à la répression par un catabolite (opérons galactose et arabinose).
En fait, pour que l’ARN polymérase puisse se fixer au site promoteur,
il faut bien entendu que le répresseur ne soit pas lié au site opérateur
mais aussi que le complexe AMP cyclique-CAP soit lié au promoteur.
La bactérie privée de source de carbone accumule l’AMP cyclique. A
l’opposé en présence de glucose, la bactérie ne possède pas suffisamment
d’AMP cyclique pour lier la CAP. Dans ce dernier cas, le complexe AMP
cyclique-CAP est en quantité insuffisante et l’ARN polymérase ne peut
pas commencer la transcription. Par contre, en présence d’une quantité
suffisante du complexe AMP cyclique-CAP sur le site promoteur, la
transcription peut se dérouler.
On sait (voir transcription chez les procaryotes) que l’ARN polymérase
se lie au niveau du promoteur par l’intermédiaire de sa sous-unité
sigma. Cette fixation de la sous-unité sigma se fait de manière
spécifique et efficace dans le cas de l’opéron lactose si le complexe
AMP cyclique-protéine CAP est fixé préalablement fixé en 5’ du
promoteur. En conclusion, le complexer AMP cyclique-CAP agit à la façon d’un régulateur positif car sa présence est requise pour l’expression génique. Le répresseur codé par le gène régulateur joue à l’opposé un rôle de régulateur négatif. Sa présence sur le site opérateur empêche la transcription des gènes de structure correspondants.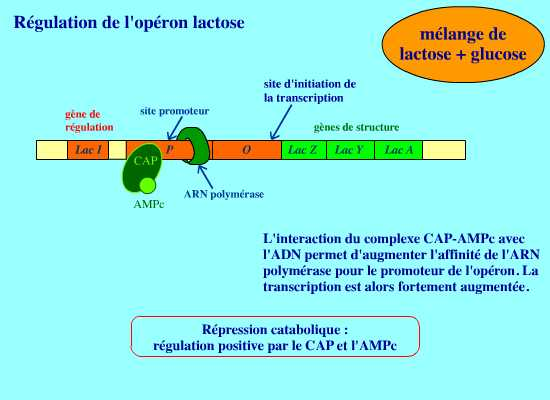
DONC :
Lorsque les bactéries, par chance, disposent en même temps de glucose et de lactose, la situation se complique !
Le répresseur de l’opéron lactose est
inactivé par l’allolactose, le site opérateur de l’opéron est donc libre
et les gènes pourraient être transcrits. Or, tant que du glucose est
présent, la bactérie va le métaboliser préférentiellement : elle n'a
donc pas besoin des enzymes nécessaires au métabolisme du lactose. Ceci
implique l'existence d'un autre mécanisme de régulation que l'on appelle
la répression catabolique. Ce n'est que lorsque la concentration en
glucose diminue que le métabolisme du lactose devient nécessaire. Un
signal de carence alimentaire est alors déclenché sous forme
d’une augmentation du taux d’AMPc. Cet AMPc forme un complexe avec la
protéine CAP (pour Catabolite gene Activator Protein, ou CRP pour cAMP
Receptor Protein). Ce complexe se lie à l’ADN en amont du site de
fixation de l’ARN polymérase. L’interaction du complexe CAP-AMPc va agir comme un inducteur et augmenter l’affinité de l’ARN polymérase pour le promoteur de l’opéron. Cette régulation positive peut permettre
d'augmenter
d’un facteur 50 la transcription de l'opéron lactose. On comprend alors
qu'en présence de glucose, il n'y a pas de complexes CAP-AMPc
disponibles : le niveau de transcription de l'opéron lactose est donc
très faible.
Conclusion L'induction de l'opéron lactose nécessite deux conditions, il faut
que le lactose soit présent et que le glucose soit absent. La
transcription de l’opéron lactose est donc sous le contrôle de deux
protéines régulatrices :
le répresseur lac-I qui se fixe au niveau de l’opérateur en absence de lactose et bloque l’ARN polymérase. C'est une régulation négative,
la protéine CAP (ou CRP) qui, sous forme d’un complexe avec l’AMPc, se
lie à l’ADN et permet d'augmenter l'affinité de l'ARN
polymérase pour le promoteur. C'est une régulation positive.
Index.
Allolactose : β-D-galactopyranosyl-(1-6)-β-D-glucopyranose.
AMPc : Adénosine 5’monophosphate cyclique, c’est un second messager intracellulaire qui est formé par l’adénylate cyclase.
ARNm polycistronique : chez les procaryotes, les ARN peuvent contenir l’information nécessaire pour former plusieurs protéines différentes.
β-galactosidase : enzyme qui permet d’hydrolyser le lactose en galactose et en glucose (elle est codée par le gène lacZ).
CAP (ou CRP) : «
Catabolite gene Activator Protein », ou CRP pour «cAMP Receptor
Protein». Cette protéine contrôle l’initiation de la transcription des
gènes du catabolisme qui va permettre d'utiliser d’autres molécules
nutritives quand le glucose est absent.
CAP-AMPc : complexe formé après l’interaction de la protéine CAP (ou CRP) avec l’AMPc.
Inducteur : molécule signal qui en se liant à une protéine régulatrice induit une augmentation de l’expression d’un gène donné.
lac A: gène de l’opéron lactose qui code pour la thiogalactoside transacétylase.
lac I: gène en amont de l’opéron lactose qui code pour le répresseur.
lac Y : gène de l’opéron lactose qui code pour la lactose perméase.
lac Z : gène de l’opéron lactose qui code pour la β-galactosidase.
Lactose : β-D-galactopyranosyl-(1-4)-β-D-glucopyranose.
Lactose perméase : Protéine membranaire qui permet l’entrée du lactose dans la bactérie (elle est codée par le gène lacY).
Opérateur : une région de l’ADN sur laquelle se fixe un répresseur pour contrôler l’expression d’un gène ou d’un groupe de gènes.
Opéron : unité d’expression génétique qui comprend un ou plusieurs gènes et des séquences régulatrices (promoteur
et opérateur) qui régule leur transcription.
Promoteur : une région d’ADN en amont du site d'initiation de la transcription sur laquelle l’ARN polymérase peut se lier.
Protéines régulatrices :
ces protéines interviennent dans le contrôle de l’expression des gènes
en se fixant sur des régions particulières de l’ADN. Elles peuvent
ainsi activer ou réprimer de façon spécifique la transcription des
gènes.
L’action de ces protéines est réversible. On parle également de facteurs de transcription.
Répresseur : protéine qui se lie sur une séquence régulatrice ou sur l’opérateur d’un gène, bloquant sa transcription.
Thiogalactoside transacétylase : cette protéine est codée par le gène lacA, son
rôle physiologique n’est pas bien connu. Elle acétyle les galactosides
non métabolisés par la β-galactosidase, cette acétylation permet ainsi
la difusion de ces sucres à travers la membrane plasmique et leur
élimation hors de la cellule.
Le système est doté d’une certaine souplesse avec répressions et
activations partielles en fonction des concentrations en glucose et/ou
lactose.Dans le cas de l’opéron lactose on dit qu’il s’agit d’un système
de type activation ( induction) : La présence même du lactose ainsi que
l’absence de glucose active le gène polycistronique et provoque
l’apparition de la batterie enzymatique propre à la séparation des
dimères de lactose en glucose et galactose.
 4.1.1.2 La répression
4.1.1.2 La répressionCas de l’opéron Tryptophane
Le tryptophane est un acide aminé
indispensable pour l’homme, ce n’est pas le cas pour les bactéries qui
peuvent le synthétiser.Pour réaliser la synthèse de cet acide aminés,il
faut que se produise tout une série de réactions toutes catalysées par
une enzyme spécifique ( 5 enzymes,5 cistrons).
Le Tryptophane est le métabolite terminal .
La synthèse de ces enzyme se réalisera quand la bactérie manque ou n’a pas de Tryptophane à disposition.
Fonctionnement en l’absence de tryptophane.
Le gène régulateur synthétise
un répresseur. Ce répresseur
se présente sous forme d’un tétramère dont les 4 sous-unités sont
identiques. Il possède une particularité essentielle. En effet,
il ne se fixe pas sur l’opérateur. On l’appelle
apo-répresseur. Ce n’est qu’en présence
d’un co-répresseur que le répresseur se fixera sur l’opérateur. En absence de répresseur
au complet (apo-répresseur et co-répresseur) fixé sur l’opérateur, l’ARN
polymérase peut se fixer sur le promoteur et commencer la
transcription.
Dans ces conditions, les gènes de structure seront
transcrits et les enzymes de synthèse du tryptophane seront
synthétisées. Le tryptophane sera produit.
Les opérons qui contrôlent la synthèse des enzymes intervenant
dans l'anabolisme fonctionnent selon le modèle de l'opéron tryptophane
présenté ci-dessous. C'est la liaison entre un aporépresseur et un
corépresseur (le produit de la chaîne de
biosynthèse) qui donne un répresseur actif. 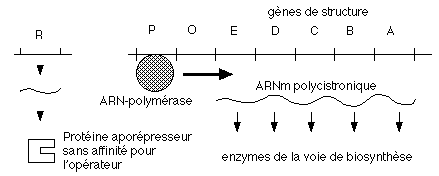 Fonctionnement en présence de tryptophane.
Fonctionnement en présence de tryptophane.
L’addition de tryptophane au milieu de culture entraîne un arrêt de la
synthèse de tryptophane. Cette synthèse est donc réprimée. Le tryptophane agissant comme un co-répresseur se lie au répresseur inactif.
Le complexe ainsi formé peut se fixer sur l’opérateur. Dans ces
conditions, l’ARN polymérase ne peut pas commencer la transcription.
Cette dernière est donc bloquée par le résultat final de l’action des
gènes de structure.En fait, le fonctionnement de l’opéron tryptophane est plus compliqué,il comporte également des séquences appelées " leader " et
" atténuateur " qui sont situées entre l’ensemble promoteur-opérateur et les gènes de structure. Lorsque la concentration en tryptophane dépasse un certain seuil,
les molécules en excès jouent le rôle de co-répresseur. La liaison apo-
et co-répresseur donne le répresseur actif qui se lie à l'opérateur. La
synthèse de tryptophane est ralentie ou stoppée.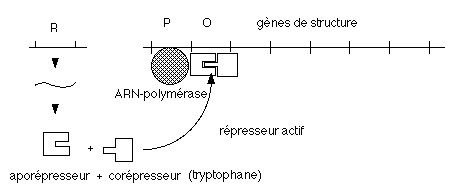 IV.1.1.3 Comparaison repression-inductionLa cellule bactérienne possède utilise donc deux types de mécanismes pour réguler la synthèse de ses protéines :
IV.1.1.3 Comparaison repression-inductionLa cellule bactérienne possède utilise donc deux types de mécanismes pour réguler la synthèse de ses protéines :1)
1er mécanisme :
répression permanenteRépression permanente qui peut être levée sur commande par un
inducteur Il s’agit d’une
dérépression par induction.
Cet exemple de régulation s’observe en général pour des enzymes qui dégradent
2)
2ème mécanisme :
synthèse permanenteSynthèse permanente, mais qui peut être arrêtée sur commande par le métabolite terminal lui même.
Ce type de régulation s’observe plutôt pour les enzymes de synthèse.
Ex : enzymes de synthèse du Tryptophane.
Ex : β galactosidase.
 IV.1.2 Régulation au niveau de la traduction
IV.1.2 Régulation au niveau de la traduction.
Cas de la synthèse des r-protéines Les r-protéines sont des protéines intervenant dans la structure des ribosomes.
Chez E.coli, le ribosome contient 52 protéines différentes qui sont donc codées par 52 gènes différents.
La fabrication des ribosomes représente pour la bactérie une dépense
d’énergie importante . Pour éviter gaspillage, le métabolisme de la
bactérie va ajuster constamment la production de ribosomes aux besoins.
1.° Les r-protéines en excès bloquent leur propre ARNmDonc une r protéine :
- Peut se fixer sur un ARNr pour former le ribosome.
- Peut se fixer sur l’ARNm pour bloquer la traduction.
2.° Affinité des r-Protéines à la fois pour ARNr et ARNmIl existe une
ressemblance structurale des sites actifs portés par ces deux ARN ; ressemblance portant sur :
- Séquence nucléotidique
- Structure secondaire, au niveau de ces sites, les ARN sont repliés
pour former une région bicaténaire ayant une forme comparable .
Donc : Concurrence entre les deux ARN pour recevoir la r-protéine
Quand les deux ARN sont disponibles, cette protéine se fixe néanmoins plus facilement sur l’ARNr.
Conclusion :
Le contrôle traductionnel est plus rapide et plus efficace que le
contrôle transcriptionnel qui ne peut avoir effet qu’après élimination
des ARNm préexistants.
IV.1.3 Nature de la reconnaissance acides nucléiques – protéinesIV.1.3.1 Exemples de reconnaissance
- Enzymes de restriction ---------- ADN
- Amino acyl ARNt synthétase ------------ ARNt
- ARN polymérase ---------- promoteur
- Répresseur ---------- opérateur
IV.1.3.2 Mécanismes possibles de la reconnaissance Jusqu’ a plus ample informé, on avance deux possibilités principales
a)
Concordance au point de vue stériqueDe nature géométrique, il y aurait concordance, harmonie entre symétrie de la protéine et de l’acide nucléique.
Exemple : la reconnaissance répresseur opérateurOpérateur : Constitué de nucléotides disposés avec une certaine symétrie ( palindrome imparfait )
Image de l’opérateur : prise à plusieurs trous.
Répresseur :
protéine, possédant une symétrie qui reconnaîtra cette séquence à
symétrie particulière comme lui étant spatialement complémentaire
Image du répresseur : prise à plusieurs broches.
b liaisons hydrogèneEntre une acide aminé et une base d’un acide nucléique, il peut exister plusieurs liaisons hydrogène.
Entre l’ adénine et le résidu 3’ asporagine, il peut s’établir deux liaisons de ce type.
Ce qui est remarquable , c’est qu’une protéine soit capable de
reconnaître une séquence de quelques nucléotides sur un acide nucléique
en comptant plusieurs millions.
IV.2 Chez les EucaryotesIV.2.1 Expression des gènesChez les eucaryotes, tous les gènes de structure ne sont pas transcrits dans toutes les cellules.
Les cellules se sont spécialisées et chaque classe de cellule exprimera différemment l’information génétique.
IV.2.2 Hypométhylation et expression des gènesQuel est le mécanisme qui permet ou empêche l’expression d’un gène.
IV.2.2.1 La méthylationUne seule base est méthylable : la
cytosine.Cette base est le plus souvent méthylée dans des séquences du type
CGCG ou encore du type CCGG . Trois % de la cytosine sont ainsi
méthylés.
IV.2.2.2 Conséquences de la méthylation de la cytosineCette méthylation de certaines séquences de gènes peut être considérée comme un
verrouillage de l’expression.
Cette conclusion provient du fait que l’on constate des schémas tels que celui qui suit :
Cellule x pas de protéine a
Le gène codant pour la protéine est méthylé.
Cellule y synthèse de protéine a
Le gène codant pour la protéine n’est pas méthylé.
Il semblerait que l’hypométhylation serait une condition déterminante
pour , nécessaire mais insuffisante pour déterminer l’expression des
gènes . Pas de règle absolue.
On avance l’explication suivante : Les radicaux méthyl empêcheraient la fixation à cet endroit des protéines nécessaires à l’expression des gènes.
IV.2.2.3 Le rôle de maintenance des ADN méthylases.
Lors de la réplication de l’ADN , ces enzymes devront méthyler certaines cytosines des deux brins fils.
Remarque :
Il est possible que la méthylation joue un rôle dans le processus de cancérogénèse.
Les cancérigènes pourraient être des inhibiteurs des ADN méthylases,
déterminant ainsi une sur expression d oncogènes par rapport aux proto
oncogènes ( taux de méthylation des oncogènes supérieur à celui des
proto oncogènes).
IV2.3 Les séquences d’ADN contrôlant l’expression des gènes.IV.2.3.1 Les enhancers ou stimulateursCes séquences activatrices agissent sur le promoteur en stimulant l’expression.
Ces séquences présentent les caractéristiques suivantes :
- Elles sont activatrices à distance des gènes ( plusieurs milliers de paires de bases).
- Elles sont situées soit en avant soit en aval du gène.
- Elles sont régulées par des facteurs diffusibles.
Remarque : il est à noter que des protéines virales sont responsables d’une activation généralisée de la transcription.
IV.2.3.2 Les silenceurs ou séquences restrictivesCes séquences ont des effets opposés à ceux des enhancers, mais elles ont les mêmes caractéristiques
V. LES MUTATIONSV.1 DéfinitionLes mutations, sont des accidents, des
erreurs qui ont lieu au cours de la
réplication de l’ADN ou de la
transcriptionV.1.1 Erreurs au cours de la réplication de l’ADNAccidents de copie :
- base mal copiée
- Base oubliée
- Base supplémentaire ajoutée
La conséquence en est la modification de l’ARNm et donc la modification de la protéine correspondante.
V.1.2 Erreur au niveau de la transcription. C’est moins grave, car chaque ADNm a une durée de vie assez courte.
V.2 Différents types de mutations V.2.1 Mutations sans changement du cadre de lecture.-
Mutations sans effets « silencieuses » : Remplacement d’un codon par un codon codant pour le même acide aminé.
Ex : UUU----------------> UUC
Phe ----------> Phe ( aa indispensable)
Aucune conséquence .
-
Mutations conservatricesCodon remplacé par un autre codon codant pour un acide aminé similaire, du même groupe.
AAA -------------> AGA
Lys ------------> Arg ( 2 aa indispensables et basiques.)
Peu de conséquences
-
Mutations faux-sensCodon remplacé par un codon donnant un acide aminé chimiquement différent
AAG GAG
Lys Glu
Acide aminé basique remplacé par un acide aminé basique : GRAVE, la protéine est modifiée,
protéine anormale.
-
Mutations portant sur un codon stopUGC -------------> UGA
Cys -------------> Stop
Particulièrement grave si cela se déroule en
début de chaîne, pas de protéine ou beaucoup plus courte.
Relativement
moins grave si en
fin de chaîne, protéine presque achevée.
L’inverse est également possible, la protéine sera alors plus longue.
V.2.2 Mutations avec changement du cadre de lecture-
La délétion : base(s) non copiée(s), manquante(s).
C’est d’autant plus grave que le déphasage se produit au début du gène
AUG/GCC/UCU/AAC/CAU/…………………..
Mét Ala Ser Asn His…………………..
AUG/- CCU/CUA/ACC/AU………..
Met Pro Leu Thr Ala………..
-
Perte d’une base.
La liaison glycosylique liant dans l’ADN les bases avec le
désoxyribose est relativement labile dans des conditions physiologiques.
A l’intérieur d’une cellule de mammifère, plusieurs milliers de bases
puriques et plusieurs centaines de bases pyrimidiques sont spontanément
perdues dans le génome d’une cellule haploïde et par jour. La perte
d’une base purique ou pyrimidique crée
un site appelé apurinique ou apyrimidique (ou site AP).-
InsertionUne base ajoutée : mêmes effets.
V.3 Exemples de conséquences de mutations.
-
Maladies des organismesEx :
la Drépanocytose, due à une modification d’une chaîne de l’hémoglobine ( remplacement d’un seul acide aminé)
Impossibilité pour cette hémoglobine de véhiculer normalement l’oxygène.
Autres maladies humaines : Mucoviscidose, Phénylcétonurie….
-
Evolution des organismesLes mutations ne sont pas toujours néfastes, il existe des mutations
qui apportent un plus ou une adaptation. Elles constituent un des
moteurs de l’évolution.
V.4 Les agents mutagènes.
Les mutations
spontanées sont
rares lors de la réplication de l’ADN.
En effet, comme nous l’avons vu, les ADN polymérases possèdent souvent une
fonction d’édition ( ADNase I et III ).
V.4.1 Les agents mutagènes chimiques-
Substances chimiques qui transforment les basesMolécules qui provoquent par exemple des désamination.
Exemple des désaminations oxydatives provoquées par HNO
2 
Mais aussi :
- Les agents alkylants. Ils entraînent l’addition de groupes alkyles aux bases (souvent des groupes -CH
3). Les agents alkylants sont
utilisés comme médicaments anticancéreux.
- Enfin,
certains agents s’intercalent entre les deux brins d’ADN,
ce sont des agents intercalants.
Ils perturbent ainsi la réplication. C’est le cas du révélateur de
l’ADN utilisé dans de nombreux laboratoires de biologie moléculaire: le
bromure d’éthydium ou B.E.T.
 V.4.2 Les agents mutagènes physiques
V.4.2 Les agents mutagènes physiquesRayons X , Ultraviolets…
L’énergie des radiations ultraviolettes est absorbée principalement
au niveau des pyrimidines de l’ADN, provoquant une excitation des
électrons et des modifications au niveau des liaisons chimiques.
- Formation de
dimères de Thymine , ce qui provoque des distorsions locales sur l’ADN, il devient impossible pour l’ADN polymérase de recopier ces deux thymines
- Formation de ponts entre les brins de l’ADN ou entre des protéines et l’ADN.
Certains agents chimiques (psoralènes par
exemple) ou physiques (UV, radiations ionisantes) peuvent conduire à des
interactions stables (« crosslinks ») entre les deux brins d’ADN. Des
interactions stables peuvent être également observées entre des
protéines et les brins d’ADN.
- Les coupures des brins d’ADN.
Les radiations ionisantes peuvent conduire à des coupures d’un brin d’ADN ou à des cassures des deux brins d’ADN.
V.5 Réparation des dimères de Thymine.V.5.1 La possibilité de réversibilité des dommages sur l’ADN.
C’est la possibilité de réparer l’ADN la plus simple, mais dans bien
des cas elle est impossible à réaliser pour des raisons cinétiques et /
ou thermodynamiques. Dans certains cas, des enzymes spécifiques comme
les photolyases peuvent transformer réversiblement un ADN lésé avec présence de dimères
pyrimidiques en un ADN normal en absorbant de la lumière. Les
photolyases sont présentes dans les bactéries, les plantes, les
champignons mais elles sont absentes chez les mammifères.
V.5.2 Correction des erreurs d’appariements.Beaucoup d’erreurs d’appariements (ou « mismatches ») sont dues à des
erreurs au cours de la réplication de l’ADN. Cependant, des erreurs
d’appariements peuvent être également produites à la suite d’une
désamination de la 5’-méthyl cytosine pour produire de la thymine qui ne
s’apparie pas correctement avec G.
Chez E. coli, la présence d’un mauvais appariement localisé est décelée par
une protéine spécifique appelée protéine MutS.
La protéine MutS reconnaît des erreurs d’appariements mais aussi des
insertions ou des délétions de quelques nucléotides. Une protéine
supplémentaire,
la protéine MutL stabilise le complexe formé entre MutS et la portion d’ADN double brin qui présente le mauvais appariement. Ce complexe va activer
une protéine MutH qui va couper le brin nouvellement synthétisé. Il est remarquable que
ce système de coupure différencie le brin parental et le brin
nouvellement synthétisé. En effet, dans l’ADN d’E. coli, les séquences
GATC sont normalement méthylées. Dans le brin nouvellement synthétisé,
la méthylation n’est pas immédiatement réalisée. La protéine MutH va
couper le brin nouvellement synthétisé à l’opposé de la séquence GATC
méthylée du brin parental. Une coopération avec la protéine uvrD
(hélicase II) est nécessaire. Le brin nouvellement formé est alors
partiellement dégradé et resynthétisé correctement par l’ADN polymérase
III. La soudure finale est assurée par l’ADN ligase.
Les organismes eucaryotes possèdent de nombreux systèmes de
réparation des erreurs d’appariements. Ainsi, certains malades atteints
d’une forme héréditaire de cancer du colon (syndrome de Lynch)
présentent des mutations dans les gènes des enzymes de réparation des
erreurs d’appariements. Ces enzymes corrigent les erreurs de réplication
apparaissant au niveau de zones de répétition de séquences
nucléotidiques courtes en tandem qui sont appelées les microsatellites.
V.5.3 la réparation par excision resynthèseCe système de réparation est présent chez les eucaryotes et les procaryotes.
Ce type de réparation est communément utilisé pour éliminer les bases
incorrectes (comme l’uracile) ou les bases transformées par des agents
alkylants. Il comprend trois étapes successives:
- Tout d’abord, une élimination de la base incorrecte par
une ADN N-glycosylase avec apparition
d’un site AP.
- Puis, une coupure du brin d’ADN
endommagé en 5’ du site AP par une endonucléase, créant ainsi un -OH
(3’) adjacent au site AP.
- Enfin, l’extension à partir de cet -OH (3’) par une ADN polymérase est réalisée avec excision du site AP.
La soudure finale est assurée par une ADN-ligase.
De nombreuses ADN N-glycosylases sont caractérisées actuellement (E.
coli). Ces enzymes sont spécifiques de tel ou tel type de lésion au
niveau d’une base lésée (par exemple: formation de la 5-méthyl
cytosine).
Nous avons vu que chez les mammifères et l’Homme, les doublets
nucléotidiques CG peuvent être méthylés au niveau de la cytosine et que
cette méthylation était en relation directe avec l’inactivation des
gènes correspondants. La désamination des cytosines méthylées aboutit à
la formation de la thymine. Une ADN glycosidase particulière reconnaît
spécifiquement le mésappriement TG et élimine T. Cependant, l’efficacité
de ce système est loin d’être parfaite. Souvent, les mutations
ponctuelles de l’ADN concernent ces cytosines méthylées, on parle de
points chauds de mutation.V.5.4 L’excision-réparation de nucléotide.
Bien que l’excision-réparation de base joue un rôle très important
comme processus de réparation de l’ADN, elle ne peut pas suffire à
corriger toutes les erreurs dans l’ADN. En particulier, la disponibilité
des ADN N-glycosylases ne peut pas être étendue à toutes les
altérations possibles des bases de l’ADN. Un autre mécanisme plus
flexible de réparation est impliqué. Ce mécanisme est appelé
excision-réparation de nucléotide. Il est disponible dans tous les organismes vivants et il implique les étapes suivantes:
- Tout d’abord, la reconnaissance du dommage sur l’ADN.
- Puis une liaison d’un complexe multi-protéique avec le site endommagé.
- Une double excision du brin endommagé suit avec coupure plusieurs nucléotides en 5’ et en 3’ de celui-ci.
- La portion oligonucléotidique contenant la lésion est dissociée entre les deux points de coupure 3’ et 5’.
- La lacune présente est comblée par une ADN polymérase.
- Une soudure finale est ensuite assurée par une ADN-ligase.
Chez E. coli, trois protéines, les produits des gènes
uvrA,
uvrB et
uvrC sont
responsables de la reconnaissance du dommage et de la coupure de l’ADN.
Le modèle actuel implique la formation initiale d’un complexe entre
deux molécules de la protéine uvrA et une molécule de la protéine uvrB
et ceci avec hydrolyse d’ATP. Ce complexe se lie avec l’ADN
préférentiellement au niveau de la zone endommagée. Il est possible que
l’ADN endommagé soit reconnu par une distorsion de la double hélice
comme c’est le cas d’une courbure de la double hélice induite par les
dimères de thymine. Après liaison de l’ADN avec la protéine uvrB, la
protéine uvrA est relâchée. Une autre protéine uvrC se lie avec la
protéine uvrB, ceci entraîne une coupure en 5’ (7 nucléotides avant le
dommage) et en 3’ (4 nucléotides après le dommage). Ces étapes demandent
la fixation de l’ATP, mais pas son hydrolyse. Une quatrième protéine
intervient, produit du gène uvrD, qui est une hélicase II, elle déplace
l’oligonucléotide endommagé. La protéine uvrB est déplacée dans l’étape
suivante quand l’ADN polymérase I (ou l’ADN polymérase II en l’absence
de l’ADN polymérase I) comble la lacune résultante. La soudure finale
est assurée par une ADN-ligase.
Un tel système de réparation existe également chez les cellules
eucaryotes. On connait d’ailleurs chez l’Homme, des anomalies génétiques
du système de réparation par exicision-réparation responsables de
maladies génétiques graves, nous citerons parmi elles, la xérodermie
pigmentaire (entraînant une sensibilité accrue aux rayons solaires et
une augmentation du risque de cancers cutanés), mais aussi le syndrome
de Bloom ou la maladie de Fanconi.
V.5.5 Les réparations post-réplicatives.
Si le système de réparation par excision-resynthèse de l’ADN est
débordé, et surtout si les zones d’ADN à réparer coincident avec la
présence de fourches de réplication, il est évident que le brin porteur
de la lésion devient impropre à servir de matrice. Dans de telles
conditions, le brin parental contiendra un dimère de thymine et l’autre
brin fils contiendra une lacune, on parle de
lacune post-réplicative. C’est une lésion grave de l’ADN.
Pour traiter de telles lésions, un système de réparation approprié va
être mis en oeuvre. Il s’agit du système des protéines Rec (pour
Recombinaison). Ce système de réparation fait donc intervenir
une recombinaison. La recombinaison correspond à un échange d’ADN ente deux segments
homologues. Cette correction fera également intervenir le système de
réparation par excision-resynthèse de l’ADN. Chez E. coli, la production
de la protéine de recombinaison appelée RecA est réduite dans les
conditions normales. Ceci est lié à une répression de la synthèse par un
répresseur protéique appelé LexA (voir contrôle de la transcription).
V.5.6 Le système S.O.S.Le système S.O.S. a été décrit initialement chez les bactéries. Il
concerne au moins une vingtaine de gènes dont les produits protéiques
sont impliqués dans les mécanismes de réparation et de recombinaison. Ce
système est remarquable parce qu’il est inductible, c’est-à-dire mis en
oeuvre par l’agression physique ou chimique. La protéine de
recombinaison RecA joue un rôle central dans un tel mécanisme de
sauvegarde. La synthèse de cette protéine est normalement réprimée par
une protéine appelée LexA ou répresseur. Si un besoin important de
protéine RecA apparaît, la répression est levée grâce à une propriété
particulière de la protéine RecA qui est capable de cliver son
répresseur ( activation de sa propriété protéolytique ).
La réponse
S.O.S se réalise en
temps très court , avec pour conséquence, une
réparation imparfaite de l’ADN :
- Non respect de la règle de complémentarité
- Suppression de la fonction d’édition.
Conclusion : Pas de blocage de la synthèse d’ADN, pas de mort cellulaire, mais apparition de mutations nombreuses.
C’est le prix de la survie
Les systèmes de réparation post réplicative et S.O.S ne sont pas mis en évidence pour le moment chezl’homme
Croquis illustrant ce chapitre



